撰稿人:吳振吉醫師
撰稿日期:2015-03-04
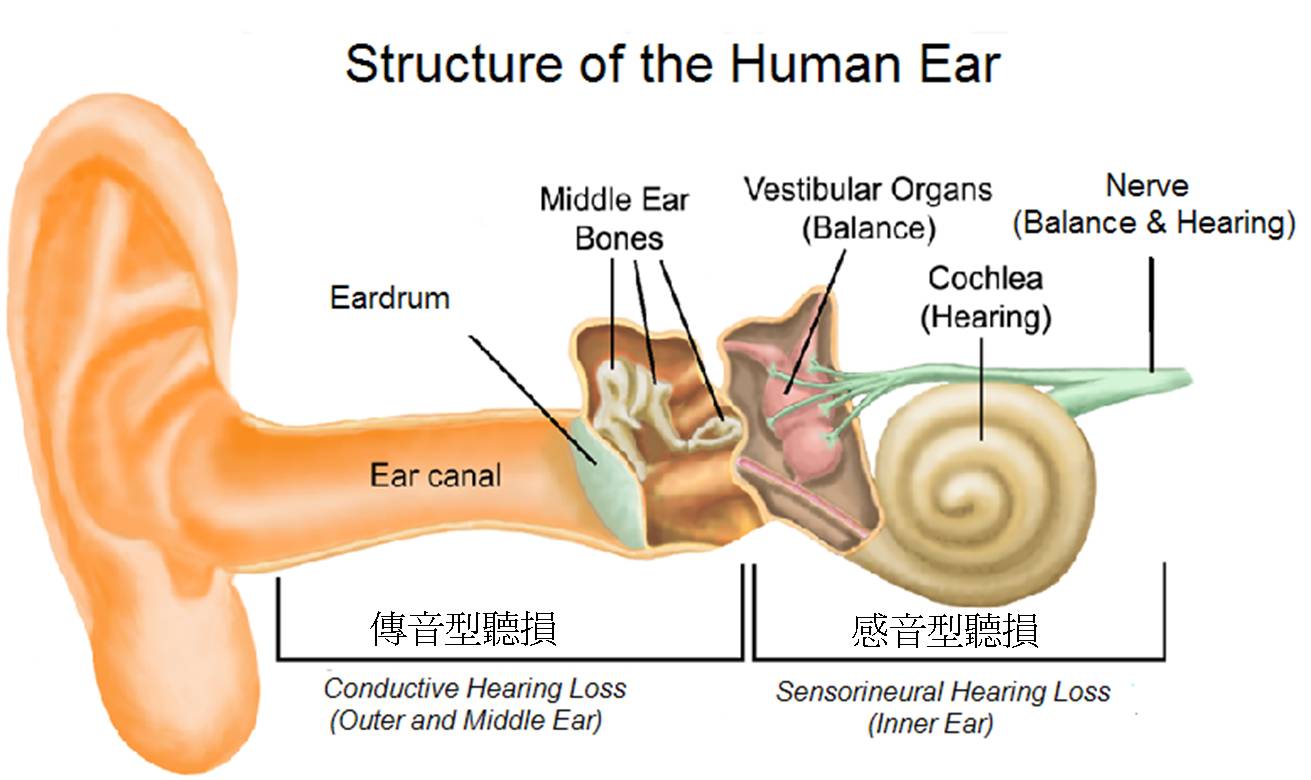
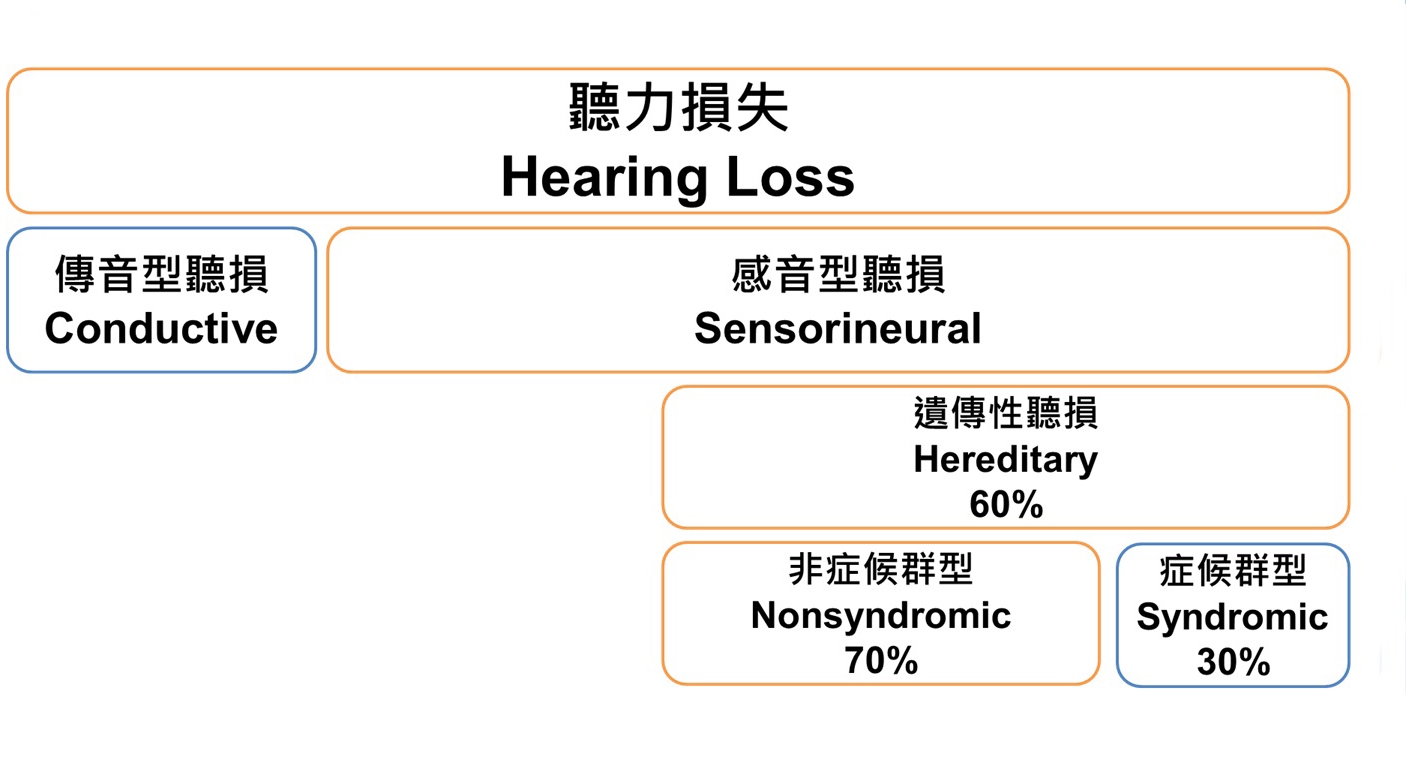
兒童聽力損失是臨床上相當常見的疾病。根據聽損發生的原因,可分為由外耳或中耳缺陷所導致的「傳音型聽損」、由內耳或聽神經缺陷所導致的「感音型聽損」,以及同時包含上述兩者的「混合型聽損」。其中,感音型聽損是人類最為常見的感覺系統缺陷。在已開發國家,每 1000 位新生兒當中,至少會有一人出現感音型聽損的症狀,隨著年齡的增長,盛行率由五歲以內幼童的2.7/1000,增加到青少年時期的3.5/1000。流行病學的研究指出,約有 60% 的感音型聽損可歸因於基因因素。此類由基因因素所造成的聽損,又稱為「遺傳性聽損」。
有30%的遺傳性聽損會伴隨著身體其他器官的異常,稱為「症候群型聽損」,而另外70%的遺傳性聽損,只出現聽力損失的症狀,稱為「非症候群型聽損」。非症候群型聽損因為缺乏可供辨識的臨床表徵,大多只能利用基因診斷的方式來進行分類。但目前已知的聽損致病基因超過100個,因此只能針對常見的基因變異去做掃描。這種極端的基因異質性,造成了診斷上的困難
遺傳性聽損大多屬於單基因的遺傳疾病。根據遺傳的模式,可分為:體染色體隱性遺傳聽損 (75~80%)、體染色體顯性遺傳聽損 (~20%),X 染色體性聯遺傳 (2~5%)、和粒線體遺傳 (~1%)。目前已經超過 100 個與聽損有關的基因,在全球的統計當中,最常見的體染色體隱性耳聾基因為 GJB2,在某些族群當中佔了將近50%的非症候群型聽損案例。
臺灣常見的耳聾基因,以 GJB2, SLC26A4 和粒線體12S rRNA三個基因為主。在我們先前的研究當中,以上三個基因變異在聽損病人當中所佔的比例依序為21.7%,14.4%,以及 3.8%。其中最為常見的變異分別為 GJB2 基因的 c.109G>A (p.V37I) 與c.235delC 變異、SLA26A4 基因的 c.919-2A>G 變異,以及粒線體 12S rRNA基因的 m.1555A>G 變異。經由三個常見基因的檢測,我們只能診斷出約三分之一的病例,另外三分之二的致病原因依然未知。但若要針對所有已知的耳聾基因去做檢測,成本會非常昂貴與耗時,在實際應用上並不可行。
近年來,次世代定序技術的出現,提供了快速、便宜且準確的基因資訊。不同於傳統直接定序一次只能針對單一基因片段,次世代定序技術以平行處理的方式,同時掃描大量基因序列,因此可快速得到超過十億個基因片段資訊。我們在 2013 年發表了第一篇針對臺灣漢人族群所做的耳聾基因次世代定序研究,並建立以次世代定序為基礎的耳聾基因檢測平台,目前的版本包括了161個已知的耳聾基因,將廣泛地應用在基礎實驗與臨床服務,進行遺傳性聽損的檢測與診斷。基因診斷的確立有助於減輕病患家屬的罪惡感與焦慮,給予完整的家族復發率諮詢,以及提供關於聽損成因與預後的關鍵資訊。
參考文獻:
- Smith, R.J., Bale, J.F., Jr., and White, K.R. (2005). Sensorineural hearing loss in children. Lancet 365, 879-890.
- Morton, C.C., and Nance, W.E. (2006). Newborn hearing screening--a silent revolution. N Engl J Med 354, 2151-2164.
- Marazita, M.L., Ploughman, L.M., Rawlings, B., Remington, E., Arnos, K.S., and Nance, W.E. (1993). Genetic epidemiological studies of early-onset deafness in the U.S. school-age population. Am J Med Genet 46, 486-491.
- Wu, C.C., Chen, P.J., Chiu, Y.H., Lu, Y.C., Wu, M.C., and Hsu, C.J. (2008). Prospective mutation screening of three common deafness genes in a large Taiwanese Cohort with idiopathic bilateral sensorineural hearing impairment reveals a difference in the results between families from hospitals and those from rehabilitation facilities. Audiol Neurootol 13, 172-181.
- Chiu, Y.H., Wu, C.C., Lu, Y.C., Chen, P.J., Lee, W.Y., Liu, A.Y., and Hsu, C.J.
- Metzker, M.L. (2010). Sequencing technologies - the next generation. Nat Rev Genet 11, 31-46.
- Brownstein, Z., Bhonker, Y., and Avraham, K.B. (2012). High-throughput sequencing to decipher the genetic heterogeneity of deafness. Genome Biol 13, 245.
- Wu, C.C., Lin, Y.H., Lu, Y.C., Chen, P.J., Yang, W.S., Hsu, C.J., and Chen, P.L. (2013). Application of massively parallel sequencing to genetic diagnosis in multiplex families with idiopathic sensorineural hearing impairment. PLoS One 8, e57369.